Haplogroupe DE
| Date d'origine | 76 500 (67 600 - 86 700) ans AP[1]. |
|---|---|
| Place d'origine | Afrique ou Asie |
| Ancêtre | CT |
| Descendants | D, E |
| Mutations définies | M1/YAP, M145 = P205, M203, P144, P153, P165, P167, P183 |
En génétique humaine, l'Haplogroupe DE est un haplogroupe du chromosome Y. Il est défini par des mutations, soit par polymorphisme nucléotidique singulier (PNS), soit par polymorphisme à évènement unique (PEU) : M1(YAP), M145(P205), M203, P144, P153, P165, P167, P183[2].
L'haplogroupe DE est souvent défini par la mutation la plus connue, dite YAP, qui est un polymorphisme à évènement unique (PEU). La mutation YAP est apparue quand une plage d'ADN appelée séquence Alu, qui se reproduit par elle-même, inséra une copie dans le chromosome Y. Un chromosome Y qui possède la mutation YAP est dit YAP-positif (YAP+), et un chromosome Y qui ne l'a pas est dit YAP-négatif (YAP-).
On estime que l'haplogroupe DE est apparu il y a environ 65 000 ans[3], ou 76 000 ans[1].
Composantes
[modifier | modifier le code]La plupart des lignages mâles DE font partie, soit de l'haplogroupe D, peut-être originaire d'Asie, la seule zone où il a été trouvé[3], soit de l'haplogroupe E, qu'on croit originaire d'Afrique de l'Est[4],[5] ou du Proche-Orient[6]. On classe le reste des lignages dans le paragroupe DE*, dont les cas confirmés sont extrêmement rares.
Distribution
[modifier | modifier le code]DE*
[modifier | modifier le code]Dans une étude mondiale de plus de 8 000 hommes comprenant 1 247 hommes du Nigeria, l'haplogroupe DE* fut observé chez seulement 5 Nigérians (5/1247)[7]. Plus récemment, un exemplaire de DE* a été trouvé chez les Nalu de Guinée-Bissau (1/17). La séquence DE* de cet individu diffère d'une mutation de la séquence DE* des Nigérians. Ceci montre un ancêtre commun, quoique la relation phylogénétique entre les deux lignages ne fut pas précisée par cette étude[8]. Une étude en 2008 détecta DE* dans deux individus du Tibet (2/594)[9].
La seule personne dans le monde à avoir été confirmée haplogroupe DE* par un test commercial est un Arabe sunnite de Damas, en Syrie[10]. Dans la base de données de yhrd.org, on trouve un haplotype similaire au sien seulement chez des Arabes de Syrie, examinés par l'étude "Donbak2006"[11] (9 sur 9 marqueurs, 1 sur 113 individus). Ceci montre que la Syrie contient un peu d'haplogroupe DE* (ce qui n'avait jamais été rapporté ailleurs qu'en Afrique de l'Ouest et qu'au Tibet par des études universitaires). Cependant, toutes les autres études universitaires faites en Syrie ont toujours échoué à trouver cet haplogroupe. Le seul haplotype en dehors de l'Afrique qui soit proche du précédent a pu être trouvé dans la base de données de smgf.org (Sorenson) et il appartient à une famille d'Arabes palestiniens de Jérusalem.
DE
[modifier | modifier le code]L'haplogroupe DE est trouvé en Afrique (haplogroupes E et DE*) et en Asie de l'Est (haplogroupes D et DE*) mais est complètement absent entre ces deux régions. La présence de DE à travers des régions largement séparées a troublé les enquêteurs qui essayent de reconstruire la migration des hommes d'Afrique vers l'Asie. À un moment, il y aurait eu une extinction des lignages DE dans les parties ouest, sud et centrale de l'Asie. Les lignages autochtones DE sont absents en Inde, une région importante dans la dispersion des humains en Asie. Cependant des lignages ont été détectés dans les populations reliques des îles Andaman[12], ce qui suggère la possibilité que des mutations délétères puissent expliquer l'extinction des lignages DE en Inde[13].

Historique
[modifier | modifier le code]L'insertion YAP fut découverte par Michael Hammer et son équipe de l'Arizona[14]. Entre 1997 et 1998 Hammer publia trois articles relatifs aux origines de l'haplogroupe DE[15],[16],[17]. Pour ces articles, l'insertion YAP se produisit en Asie. Plus récemment en 2007, des études comme celle de Chandrasekar et al. 2007 citent les publications de Hammer en prenant aussi parti pour une origine asiatique de l'insertion YAP[6].
Les scénarios développés par Hammer comprennent une sortie d'Afrique il y a plus de 100 000 ans, l'insertion YAP+ sur un chromosome Y asiatique il y a 55 000 ans et une migration de retour du YAP+ de l'Asie vers l'Afrique il y a 31 000 ans par son sous-clade, l'haplogroupe E[17]. Cette analyse était basée sur le fait que les plus vieux lignages africains, comme les haplogroupes A et B, étaient YAP négatifs tandis que le plus jeune lignage, l'haplogroupe E était YAP positif. l'haplogroupe D, qui est YAP positif, était apparemment un lignage asiatique car étant seulement trouvé en Asie de l'Est, avec des fréquences élevées au Japon et au Tibet. Parce que des mutations qui définissaient l'haplogroupe E furent observées déjà présentes dans un état ancestral de l'haplogroupe D, et parce que l'haplogroupe D ayant 55 000 ans est considérablement plus vieux que l'haplogroupe E avec 31 000 ans, Hammer en déduisit que l'haplogroupe E était un sous-clade de l'haplogroupe D[17].
En 2000, nombre de scientifiques avaient commencé à remettre en question l'hypothèse d'une origine asiatique de l'insertion YAP. Underhill et al. 2000 identifièrent la mutation D-M174 qui définit l'haplogroupe D. L'allèle M174 n'a été trouvé dans aucun lignage africain comme l'haplogroupe E. La découverte de M174 signifiait que l'haplogroupe E ne pouvait pas être un sous-clade de l'haplogroupe D. Cette découverte contredisait l'idée d'une origine asiatique de YAP+, basée sur les mutations M40 et M96 qui définissent l'haplogroup E selon Underhill et al. 2000. La seule existence de M174 démontrait à elle seule une origine africaine de l'insertion YAP[18].
D'autres arguments furent apportés soutenant l'origine africaine de YAP dans Underhill et al. 2001. Ces arguments sont[4]:
- L'Afrique a une plus grande fréquence de YAP (> 80 %). Tandis que YAP+ en Asie a une distribution géographique assez restreinte, surtout à des fréquences basses et modérées (moyenne 9,6 %) en Asie de l'Est[9].
- On a relevé qu'il n'y a pas de preuve archéologique de migration retour vers l'Afrique, et qu'il n'y a pas jusqu'ici de preuve solide de migration retour vers l'Afrique de l'ADN Y, de l'ADN mitochondrial ou de l'ADN autosomal[4].
- Quoique l'haplogroupe C semble originaire d'Asie à la même époque que l'origine de l'haplogroup DE, l'haplogroupe C ne montre aucun signe de migration retour vers l'Afrique.
L'origine africaine de YAP+ est aussi soutenue par des études sur l'haplogroupe E. Dans Altheide et Hammer 1997, ces auteurs soutiennent que l'haplogroupe E serait apparu en Asie sur un allèle ancestral YAP+, avant de faire une migration retour vers l'Afrique[16]. Cependant quelques études, comme celle de Semino et al., indiquent que les fréquences et diversités les plus élevées de l'haplogroupe E sont en Afrique, l'endroit le plus probable d'origine de l'haplogroupe E[9],[19].
Les modèles soutenant une origine africaine ou une origine asiatique de l'insertion YAP+ requièrent tous deux l'extinction du chromosome ancestral YAP pour expliquer la distribution actuelle du polymorphisme YAP+. Le paragroupe DE* ne possède ni les mutations qui définissent l'haplogroupe D, ni celles pour l'haplogroupe E. Si le paragroupe DE* avait été trouvé sur un continent et non sur l'autre, cela aurait privilégié une théorie par rapport à l'autre[20].
L'haplogroupe DE* a récemment été trouvé au Nigeria[7], en Guinée-Bissau[8] et aussi au Tibet[9]. La relation phylogénétique entre les trois séquences DE* doit être encore établie, mais on sait que les séquences en Guinée-Bissau diffèrent des séquences nigérianes par au moins une mutation[8]. Weale et al. soutiennent que la découverte de DE* parmi les Nigérians recule la date du dernier ancêtre commun des chromosomes YAP africains. Dans leur opinion, ceci a l'effet de réduire la fenêtre temps à travers laquelle une possible migration retour d'Asie vers l'Afrique pourrait s'être produite[7].
Chandrasekhar et al. 2007, ont soutenu l'origine asiatique de YAP+. Ils affirment :
« La présence de l'insertion YAP dans les tribus du nord-est de l'Inde et chez les insulaires des îles Andaman avec l'haplogroupe D suggère qu'est apparu dans un chromosome ayant le marqueur M168 (haplogroupe CT), l'insertion YAP et la mutation M174 définissant l'haplogroupe D en Asie du Sud. »
Ils soutiennent aussi que les porteurs de YAP+ retournèrent en Afrique avec d'autres haplogroupes eurasiens. Parmi ceux-ci, l'haplogroupe R1b1* (18 000 - 23 000 ans), observé avec une fréquence spécialement élevée dans quelques tribus du Nord Cameroun, et l'haplogroupe T (25 000 - 30 000 ans), observé à des fréquences peu élevées en Afrique. Mais l'haplogroupe E avec un âge de 50 000 ans est beaucoup plus vieux que ces haplogroupes et a été observé avec des fréquencees de 80 - 92 % dans une bonne partie de l'Afrique.
Dans un article de presse sur une étude de Karafet et al. (2008), Michael Hammer a révisé les dates d'origine de l'haplogroupe DE de 55 000 ans d'âge à 65 000 ans. Pour l'haplogroupe E, Hammer a révisé les dates d'apparition de 31 000 ans d'âge à 50 000 ans. Hammer est aussi cité pour avoir dit : « L'âge de l'haplogroupe DE est d'environ 65 000 ans, juste un peu plus récent que l'autre lignage majeur à avoir quitté l'Afrique, qui est donné pour avoir environ 70 000 ans d'âge », par lequel il admet que l'haplogroupe DE quitta l'Afrique avec l'haplogroupe CF[21].
Peter Underhill soutient qu'il y aura toujours une incertitude sur les origines précises des variants de séquences ADN comme YAP à cause du manque de connaissance concernant la démographie préhistorique et les mouvements de populations. Cependant Underhill prétend que, avec toute l'information disponible, l'hypothèse de l'origine africaine du polymorphisme YAP+ est plus parcimonieuse et plus plausible que celle de l'origine asiatique[13]. D'autres auteurs ont publié ou copublié des travaux soutenant l'origine africaine de YAP+, parmi lesquels Luigi Luca Cavalli-Sforza[18], Toomas Kivisild[13], Spencer Wells[20], Linda Stone et Paul F. Lurquin[22].
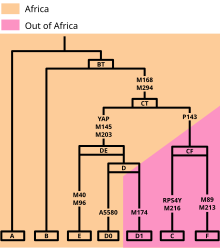
Arbre
[modifier | modifier le code]Cet arbre phylogénétique de l'haplogroupe DE est basé sur l'arbre YCC 2008[3] et les recherches publiées s'y rapportant.
- DE (M1/YAP, M145, P205, M203, P144, P153, P165, P167, P183)
Références
[modifier | modifier le code]- (en) Haber M, Jones AL, Connel BA, Asan, Arciero E, Huanming Y, Thomas MG, Xue Y, Tyler-Smith C, « A Rare Deep-Rooting D0 African Y-chromosomal Haplogroup and its Implications for the Expansion of Modern Humans Out of Africa », Genetics, (PMID 31196864, DOI 10.1534/genetics.119.302368), genetics.302368.2019
- page ISOGG
- (en) Karafet TM, Mendez FL, Meilerman MB, Underhill PA, Zegura SL, Hammer MF, « New binary polymorphisms reshape and increase resolution of the human Y chromosomal haplogroup tree », Genome Research, vol. 18, no 5, , p. 830–8 (PMID 18385274, PMCID 2336805, DOI 10.1101/gr.7172008, lire en ligne)
- (en) Underhill, Genetic, Linguistic and Archaeological Perspectives on Human Diversity in Southeast Asia, New Jersey, World Scientific, , 172 p. (ISBN 978-981-02-4784-3, lire en ligne), « The case for an African rather than an Asian origin of the human Y-chromosome YAP insertion »
- (en) Cruciani F, La Fratta R, Santolamazza P, et al., « Phylogeographic analysis of haplogroup E3b (E-M215) y chromosomes reveals multiple migratory events within and out of Africa », Am. J. Hum. Genet., vol. 74, no 5, , p. 1014–22 (PMID 15042509, PMCID 1181964, DOI 10.1086/386294, lire en ligne)
- (en) Chandrasekar A, Saheb SY, Gangopadyaya P, et al., « YAP insertion signature in South Asia », Ann. Hum. Biol., vol. 34, no 5, , p. 582–6 (PMID 17786594, DOI 10.1080/03014460701556262, lire en ligne)
- (en) Weale ME, Shah T, Jones AL, et al., « Rare deep-rooting Y chromosome lineages in humans: Lessons for Phylogeography », Genetics, vol. 165, no 1, , p. 229–34 (PMID 14504230, PMCID 1462739, lire en ligne)
- (en) Rosa A, Ornelas C, Jobling MA, Brehm A, Villems R, « Y-chromosomal diversity in the population of Guinea-Bissau: a multiethnic perspective », BMC Evol. Biol., vol. 7, , p. 124 (PMID 17662131, PMCID 1976131, DOI 10.1186/1471-2148-7-124, lire en ligne)
- (en) Shi H, Zhong H, Peng Y, et al., « Y chromosome evidence of earliest modern human settlement in East Asia and multiple origins of Tibetan and Japanese populations », BMC Biol., vol. 6, , p. 45 (PMID 18959782, PMCID 2605740, DOI 10.1186/1741-7007-6-45, lire en ligne)
- « Sidoroff (Sidorov) », sur familytreedna.com (consulté le ).
- « springerlink.com/content/qt72w… »(Archive.org • Wikiwix • Archive.is • Google • Que faire ?).
- Underhill et al. 2007
- (en) Underhill PA, Kivisild T, « Use of y chromosome and mitochondrial DNA population structure in tracing human migrations », Annu. Rev. Genet., vol. 41, , p. 539–64 (PMID 18076332, DOI 10.1146/annurev.genet.41.110306.130407, lire en ligne)
- (en) Hammer MF, « A recent insertion of an alu element on the Y chromosome is a useful marker for human population studies », Mol. Biol. Evol., vol. 11, no 5, , p. 749–61 (PMID 7968488, lire en ligne)
- (en) Hammer MF, Karafet T, Rasanayagam A, et al., « Out of Africa and back again: nested cladistic analysis of human Y chromosome variation », Mol. Biol. Evol., vol. 15, no 4, , p. 427–41 (PMID 9549093, lire en ligne)
- (en) Altheide TK, Hammer MF, « Evidence for a possible Asian origin of YAP+ Y chromosomes », Am. J. Hum. Genet., vol. 61, no 2, , p. 462–6 (PMID 9311756, PMCID 1715891, DOI 10.1016/S0002-9297(07)64077-4, lire en ligne)
- (en) Hammer MF, Spurdle AB, Karafet T, et al., « The geographic distribution of human Y chromosome variation », Genetics, vol. 145, no 3, , p. 787–805 (PMID 9055088, PMCID 1207863, lire en ligne)
- (en) Underhill et al, « The phylogeography of Y chromosome binary haplotypes and the origins of modern human populations », Ann. Hum. Genet., (lire en ligne) [PDF]
- (en) Semino O, Magri C, Benuzzi G, et al., « Origin, diffusion, and differentiation of Y-chromosome haplogroups E and J: inferences on the neolithization of Europe and later migratory events in the Mediterranean area », Am. J. Hum. Genet., vol. 74, no 5, , p. 1023–34 (PMID 15069642, PMCID 1181965, DOI 10.1086/386295, lire en ligne)
- (en) Wells RS, Yuldasheva N, Ruzibakiev R, et al., « The Eurasian heartland: a continental perspective on Y-chromosome diversity », Proc. Natl. Acad. Sci. U.S.A., vol. 98, no 18, , p. 10244–9 (PMID 11526236, PMCID 56946, DOI 10.1073/pnas.171305098, lire en ligne)
- Scientists reshape Y chromosome haplogroup tree gaining new insights into human ancestry
- (en) Linda Stone, Genes, Culture, and Human Evolution, Malden, MA, Blackwell, , 314 p., relié (ISBN 978-1-4051-5089-7, lire en ligne), « Voyages, Prehistoric Human Expansions », p. 185
|
Haplogroupes du chromosome Y (Y-ADN) | ||||||||||||||||||||||||
| Plus récent ancêtre patrilinéaire commun | ||||||||||||||||||||||||
| A | ||||||||||||||||||||||||
| BT | ||||||||||||||||||||||||
| B | CT | |||||||||||||||||||||||
| DE | CF | |||||||||||||||||||||||
| D | E | C | F | |||||||||||||||||||||
| G | H | IJK | ||||||||||||||||||||||
| IJ | K | |||||||||||||||||||||||
| I | J | LT | K2 | |||||||||||||||||||||
| I1 | L | T | MS | P | NO | |||||||||||||||||||
| M | S | Q | R | N | O | |||||||||||||||||||
| R1 | R2 | |||||||||||||||||||||||
| R1a | R1b | |||||||||||||||||||||||